
Maxime ARQUE
Coordonnées
Thèmes de recherche
PhD project : Pangenome dynamics and functional sharing within aquatic microbial communities
Microbial ecosystems, whether terrestrial, marine, or freshwater, exhibit ecological and evolutionary characteristics shaped by their specific environments. Soils, which are heterogeneous and highly unstable environments, harbor microorganisms with large gene repertoires that confer a strong adaptive potential to cope with highly variable physicochemical conditions.
Aquatic ecosystems, both marine and freshwater, are comparatively more stable and homogeneous, yet they display pronounced trophic gradients ranging from nutrient-rich (eutrophic) to nutrient-poor (oligotrophic) environments. These gradients impose strong selective pressures that profoundly influence the structure and functioning of microbial communities. In oligotrophic environments, bacteria with small cell volumes and reduced genome sizes are frequently observed. This is notably the case for the marine genera Ca. Pelagibacterales (SAR11) and Prochlorococcus, as well as the freshwater Nanopelagicales (acI clade). These traits are commonly interpreted as a genome streamlining strategy, aimed at minimizing the energetic costs associated with genome replication and maintenance, while optimizing nutrient uptake through an increased surface-to-volume ratio.
Genome streamlining is characterized by several genomic signatures, including reduced GC content, a low proportion of pseudogenes, and a decrease in gene redundancy associated with an overall simplification of metabolism. These features reflect strong purifying selection against non-essential functions and the accumulation of neutral sequences. Genome reduction is often accompanied by auxotrophies, that is, the inability to synthesize certain compounds. In such cases, metabolic functions are either compensated by compounds readily available in the environment or effectively “outsourced” to other members of the community, in accordance with the Black Queen Hypothesis (BQH). This evolutionary hypothesis posits that the loss of costly functions can become advantageous when these functions are provided by co-occurring organisms within the ecosystem (fig. 1). However, the context and consequences of genome reduction, particularly in relation to the BQH, remain poorly understood. While moderate genome reduction may enhance energetic efficiency and growth in stable, nutrient-poor environments, excessive functional losses can limit metabolic plasticity, potentially reducing fitness and increasing vulnerability to environmental fluctuations.
In this context, this doctoral project aims to disentangle the evolutionary mechanisms underlying genome reduction in freshwater Actinobacteria by assessing the respective roles of genome streamlining and metabolic dependency. Using a comparative bioinformatics framework based on existing genomic and metagenomic datasets, the project will analyze patterns of gene gain and loss, functional repertoire dynamics, and the emergence of auxotrophies to better understand how metabolic functions are lost, maintained, or shared within aquatic microbial communities.
Keywords: Actinobacteria, aquatic ecosystems, streamlining, Black Queen Hypothesis (BQH), auxotrophies
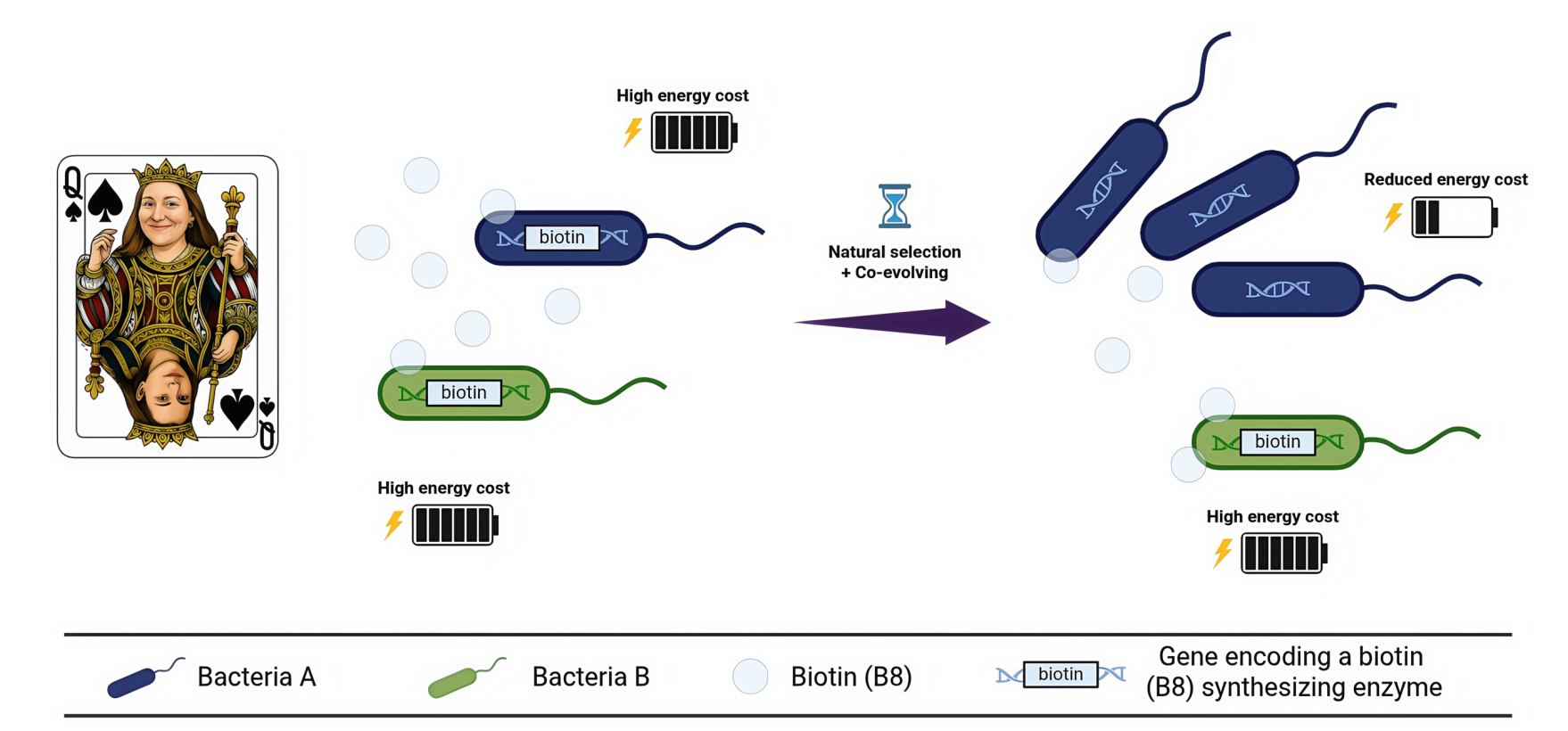
Figure 1 : The Black Queen Hypothesis is an evolutionary framework proposing that some species can lose energetically costly biological functions when these functions are reliably provided by other members of the community (Morris, 2012). In this scenario, gene loss is not inherently detrimental, rather, it can represent an adaptive strategy to reduce metabolic costs, as long as the lost function remains essential and is maintained by selection acting on function-retaining organisms (Hesse & O’Brien, 2024). This process leads to metabolic interdependencies, in which certain taxa retain and express so-called "leaky" functions that incidentally benefit other community members, thereby facilitating genome streamlining without compromising access to essential resources.
For example, consider two co-occurring bacterial species that initially both harbor the complete biosynthetic pathway for biotin, a metabolically costly vitamin to produce. Under sustained co-evolution and selective pressure, a division of labor may emerge within the community. If one species maintains or upregulates biotin biosynthesis and releases sufficient amounts into the environment, the other species may gain a selective advantage by losing part or all of the corresponding biosynthetic genes. This gene loss reduces genomic and metabolic costs while preserving access to an essential cofactor through environmental uptake. In this context, biotin auxotrophy represents an evolutionary strategy of economization, in which gene loss is favored because the function is effectively supplied by the surrounding microbial community, tightly linking individual fitness to community structure and metabolic interdependencies.

